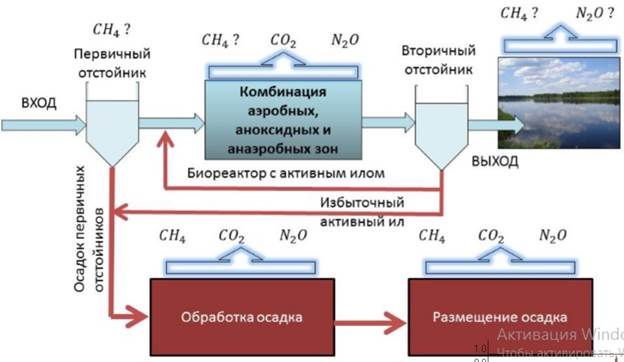
Гибридизация
ДНК-маркировки с использованием флуоресцентных красителей позволяет в отличие от радиоизотопных
Метки мгновенную оценку результатов Гибридизации в разделе "real time"
Условия. Как уже на примере РЫБА в 3.1 показано, можно также в чип
Гибридизация путем одновременного использования нескольких флуоресцентных красителей
различные вопросы будут даны ответы в эксперименте. Чаще всего происходит
установка флуоресценции красителей на фермент вносили в пути PCR, ник-
Translation или "random primed labelling" путем добавления Нуклеотидов, в которых
это Fluorophor связаны через линкер ковалентно.
Выделение ДНК во время ПЦР-реакции требует высокой долей
gelabelten Нуклеотидов. Как 3.6.1 явствует, содержит amplifi украсила только ДНК
соотношении gelabelten к ungelabelten Нуклеотидов от 1:4 примерно на каждый
сотый знаний отметка. Теоретически соотношение 30% было бы в случае G+C-
4. Обсуждение
_____________________________________________________________________________________________________________________________________________________________________________________
Lactococcus каждая 25. Base это Fluorophor носить. Из этого можно сделать вывод, что установка
флюоресценции меченных dNTPs благодаря используемым в ПЦР ДНК-полимеразы сильно
будет дискриминирован. ДНК-маркер по ПЦР-реакция появляется в сравнении
на выход скорее такие запросы не являются эффективными и сравнительно дорого.
Для выделения геномной ДНК из Закваски или брожения образцы
exudes на специфически связывающие грунтовки на основе метода. При ник-
Translation или "random primed labelling" флуоресцирования отмечены нуклеотиды в
ДНК с неизвестной последовательностью встроенный. Как Рис. 30 показывает, можно с помощью этих методов
Маркировка ставки в среднем на 25-30 молекул красителя на 1000 Нуклеотидов
быть достигнуто. Эти методы для внутренних Labelling неизвестных последовательностей ДНК
теперь найти широкое применение, например, в области медицины при доказывании
герпес симплекс вирус (Broketa et al., 2001), при анализе точечных мутаций
(Iwahana et al., 1996) или при сравнении генома Hybridisierungen (Rosenberg et al.,
1998).
Другая, также ферментативные Labelling метод через закрывающий тег
ДНК-фрагментов с терминальной Polynukleotid-трансферазы (Kodym и
Horth, 1995; Woynarowska et al., 2000). Преимущество этого метода основан на непосредственном
Quantifizierbarkeit сигнала, поскольку каждый гибрид начал фрагмент раздел
Флуоресценция испускает сигнал. С другой стороны, система оставляет себе это не доказательство
Повышение чувствительности добиться только при гибридизации низкое количество ДНК,
как для выявления патогенов в Закваски или брожения образцов,
играют жизненно важную роль.
Помимо описанных выше ферментативные методы Маркировки химических стоят
Labelling-процедуры, при которых, например, псорален-биотин, Тетра метил родамина-Isothiocyanat,
Флуоресцеин Isothiocyanat, Тетра метил родамина-succinimide эфир или цис-производные платины
используются, доступны (Proudnikov и Mirzabekov, 1996; Saffran et al., 1988;
Wiegant et al., 1999). Преимуществами этих методов являются их применимость на ДНК и
РНК, хорошей сочетаемости с для гибридизации на ДНК-чипе необходимые
Фрагментация и последнее, но не менее низкой стоимости. Так, цены на который составляют
ферментативный отметка 1 мкг геномной ДНК путем встраивания Нуклеотидов в
ПЦР-реакция на US$ 50.00, через "random primed labelling" на US$ 15.00 и через
Endlabelling с терминальной трансферазы на US$25.00, в то время как при химической
Метки псорален-биотин только затраты в размере US$ 0.5 возникнуть (Zhang et
al., 2001).
4. Обсуждение
_____________________________________________________________________________________________________________________________________________________________________________________
Вид
Нынешние ДНК-аналитические методы позволяют даже при значительной методических
Усилий только один снимок из исходной культуры или бродильной пробы. Которые
Образование ДНК-последовательностей других бродильные бактерии и уместное
Развитие ДНК-чип-технологии впервые предлагаем возможность на все
Производство молочных продуктов, участвующих организмов и генов в одном эксперименте
захватить и их, чтобы Произойти в различных стадиях Производства друг с другом
сравнить (Kuipers, 2001).
5. Резюме
_____________________________________________________________________________________________________________________________________________________________________________________
Резюме
Предметом этой работы является молекулярно-генетической характеристике является
Молочнокислые бактерии и фаги в Творог брожений с нормальной или нарушенной
Мрачной истории.
Благодаря флуоресцентной in situ гибридизации целых клеток бактерий с против rRNA
направленных зондов мог быть исключена изначально, что разлад в Скисанием
нестабильной Творог брожений брожения на нетипичные бактерии, среди которых
Патогены из родов Salmonella, Staphylococcus, Campylobacter или листерии
может находиться, это вызвано. Снижение окисление идет с сильным
Сокращение "метаболически активные" клетки вместе. Показывает начала ферментации
Анализ состава Популяции есть 98% - ной доли L. lactis ssp. cremoris
от общего количества клеток. На всех стадиях Брожения, доля двух других меняет
на ферментации микроорганизмов, участвующих L. lactis ssp. lactis и Leuconostoc
mesenteroides лишь незначительно, в то время как на момент раздражающий количество отказа
Подвид L. lactis ssp. cremoris примерно на 1% падает.
Для характеристики генетического потенциала на творог брожения участников
Организмы были 7 стартовых культур, 282 L. lactis ssp. cremoris -изоляты, которые
в разное время из брожений стартовая культура Probat с нормальной 8/0
или нарушенным скисанием были приняты, а также четыре в продаже
Пищу творог образцы в отношении мастер-Плазмиды-кодированных фаги гены устойчивости
изучены. Из-за широкого ген спектра до 17 различных генов,
самые подавлять сорбцию фаги или инъекции ДНК, через эти ограничения
безвредно или сделать сборку фаги протеины предотвращают, кажется
Стартовых культур хорошие защитные активы фаги против различных видов
владеть. Непрерывное окисление возникает, несмотря на фаги инвазии, когда
Микроорганизмы четыре или больше несут гены устойчивости. При Фаги обороноспособность
Механизмы резистентности предпочел деградировать фаги независимо от вида ДНК.
Количество генов резистентности снижается за счет потери Плазмиды бактериофаги могут
Исходной культуры бактерий lysieren. За счет минимизации числа на брожение
вовлеченных клеток, снижается активность Окисление или поставляется полностью
Изнемогают.
Последовательность анализа доказанных содержит гены, как правило, 95-98%
Последовательность идентификаторов в базах данных за возложили последовательности. В одном в торговле
доступные пищу творог был в дополнение к известным из литературы последовательность
5. Резюме
_____________________________________________________________________________________________________________________________________________________________________________________
abiP -разновидность гена abiP определены, их кодирующий последовательность "в рамке"
Вставка с 390 Нуклеотидов несет.
Анализ дополнительных Плазмид-картой гены, отвечающие за образование лактата во время
Окисление или появления вкусовых компонентов, таких как диацетил и Acetoin
из цитрат удаление несут ответственность, аналогичная картина выявляются как следствие
Фаги генов резистентности. Также здесь число бактерий в ячейках принимает
генетические маркеры при брожений с нарушениями окисление с поведением.
Характеристика брожений в содержащихся фаги вида показывает запись
из P335-фаги через стартовые культуры предположительно в lysogenem состояние и
Загрязнения с облигатным литические фаги 936 вида о молоке или о
Остатки от производственного оборудования.
P482, несколько изолированный представители этих фаги вида, полностью секвенировали.
Это 30945 bp длинный геном разделен на 55 ORFs, которые в ранней, средней и поздней
Транскрипционные единицы организованы. Несмотря на высокое Сходство с последовательностью записей в
базы данных, могли быть назначены только 16 генов функции. Были выявлены
субъединиц для Terminase, возможных протеинов, формирующих структуру фаги,
Holin и лизин, субъединиц ДНК-полимеразы и HNH-Endonukleasen. В
P482-специфические ORF 11, мол на хозяин специфичности, ORF 13 при строительстве
Фаги хвоста участвуют. Характерные аминокислотные мотивы только в P482
выявленные ORFs 32 оставить Метилтрансферазы деятельности предполагают. Как пример
из ORFs 11, 31, 37 или 50 показывает, возникает разнообразие Lactococcus -фаги меньше
за счет точечных мутаций, но и, вероятно, вызвано в Модульной теории, описанной
Обмен целых генов или Genabschnitte. Появление фаги вариант
P482∆ORF11, делеция гена ORFs 11 отсутствует, а также указание на то.
Общий учет всех этих параметров в эксперименте предлагает чип-
Гибридизация интересную перспективу будущего. Необходимые эксперименты
для выделения геномной начало культуры-ДНК с целью введение ферментативных
одним флуоресценции красителя в Nick-Translation или "random primed labelling"
средняя Монтажа обеспечили баз расчета одна Fluorophor за 30-40, но
для повседневного использования слишком дорого. Поэтому необходимо методы в будущем химической Маркировки
быть прибегают.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
Литература
1. Alatossava, T. и T. R. Klaenhammer (1991) Molecular characterization of three small
изометрия-headed bacteriophages which vary in their sensitivity to the lactococcal фага
resistance плазмиды pTR2030. Заявл. Environ. Microbiol. 57: 1346-1353.
2. Эллисон, G. E. и T. R. Klaenhammer (1998) фага resistance mechanisms in lactic acid
bacteria. Int. J. Dairy 8: 207-226.
3. Эллисон, G. E., D. Angeles, н. Тран-Динь и н. к. Верма (2002) Complete genomic
sequence of SfV, a serotype-converting temperate of bacteriophage шигеллы Флекснера. J.
Bacteriol. 184: 1974-1987.
4. Amann, R. I., W. Ludwig и K.-H. шлифмашины (1995) identification and Phylogenetic в
situ detection of individual microbial cells without выращивание грибов. Microbiol. Преподобный 59: 143-
169.
5. Amann, р., ф. о. горбун и А. Neef (1997) Modern methods в подповерхностные
microbiology - in situ identification of microorganisms with nucleic acid probes. FEMS
Microbiol. Апок. 20: 191-200.
6. Anba, J., E. Bidnenko, А. Хиллер, д. Честно и M. C. Шопена (1995) Characterization
of the lactococcal abiD1 гены coding for фага абортивных infection. J. Bacteriol. 177:
3818-3823.
7. Андресен, А., А. Гайс, & Krusch и тойбер м. (1984) Плазмиды profiles of mesophilic
dairy starter cultures. Молоко наук 39: 140-143.
8. Баба, Т. Ф. Takeuchi, М. Курода, H. Юдзаве, К. Аоки, А. Огучи, Ю. Нагаи, Ивама Н.,
К. Асано, т. к. Наими, х. Курода, л. Кюи, к. Ямамото и Hiramatsu K. (2002) геномы
and virulence determinants of high virulence community-acquired Staphylococcus
aureus MRSA. Lancet 359: 1819-1827.
9. Baranyi, U., R. Klein, W. г-н любитц, т. е. Крюгер и А. Витте (2000) The archaeal
halophilic virus-encoded ДЭМ -like метилтрансферазы M. phiChi-I methylates adenine
residues and дополняет ДЭМ mutants in the low salt environment of Escherichia coli.
Мол. Microbiol. 35: 1168-1179.
10. Basaran, п., н. и Basaran и. Cakir (2001) Molecular differentiation of Lactococcus
lactis ssp. lactis and cremoris strains by ribotyping and site specific PCR. Current
Microbiol. 42: 45-48.
11. Beck, C., S. "Кранц", м. Solmaz, M. Roth и А. Jeltsch (2001) How does a ДНК
interacting ферменты change its specificity during molecular evolution? A site-directed
mutagenesis study at the DNA binding site of the ДНК(Adenine-N6)-methyltransferase
Eco RV. Biochemistry 40: 10956-10965.
12. Бек, H. (2002) Обзор патогенные микроорганизмы в пищевых продуктах. Лекция
на конференции "молекулярно-биологические методы обнаружения для патогенетического
Микроорганизмы" Баварского Landesamts для здоровья и еда
безопасности, обершлайсхайм, 1./2. Июль.
13. Беккер, А. и м. Gold (1988) Prediction of АТФ reactive center in the small subunit,
gpNu1, of the фага лямбда terminase ферментов. J. Mol. Biol. 139: 219-222.
14. Beimfohr, с. А. Краузе, Amann R., W. Ludwig и K. H. Sander (1993) In Situ
identification of Lactococci, Enterococci и Streptococci. Syst. Заявл. Microbiol. 16:
450-456.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
15. Beimfohr, C. W. Ludwig и K. H. Шлейфер (1997) Рапид genotypic of differentiation
Lactococcus lactis subspecies and биовар. Syst. Заявл. Microbiol. 20: 216-221.
16. Bekal-si Али, с., с. Divies Prevost и H. (1999) organization of the Genetic citCDEF
locus and identification of mae and clyR from genes Leuconostoc mesenteroides. J.
Bacteriol. 181: 4411-4416.
17. Benson, K. H., J. J. ГОДО, р. Renault, H. G. Гриффин и м. Ю. Gasson (1996) Effect of
ilvBN -encoded α-acetolactate synthase expression on диацетил production в Lactococcus
lactis. Заявл. Microbiol. Biotechnol. 45: 107-111.
18. Бересфорд, T. P. J., L. Ward и J. H. A. W. Jarvis (1993) regulated Temporally
transcriptional expression of the genomes of lactococcal bacteriophages с2 и sk1. Заявл.
Environ. Microbiol. 59: 3708-3712.
19. Betzl, D., W. Ludwig и K. шлифовальная машина (1990) Identification of and lactococci
enterococci by colony hybridization with 23S рРНК-targeted олигонуклеотидных зондов.
Заявл. Environ. Microbiol. 56: 2927-2929.
20. Би, X. и л. ф. Liu (1994) RecA - независимый и recA -dependant intramolecular
плазмиды recombination. Дифференциальная homology requirement and distance effect. J. Mol.
Biol. 235: 414-423.
21. Би, X. и л. ф. Лю (1996) RecA -независимый DNA recombination between повторяющиеся
sequences: mechanisms and тип. Proc. Natl. Acad. Sci. США 54: 253-292.
22. Bidnenko, е., д. и Честно M. C. Шопена (1995) фага operon involved in sensitivity
to the Lactococcus lactis абортивных infection mechanism abiD1. J. Bacteriol. 177: 3824-
3829.
23. Bidnenko, е., с. Мерсье, Ж. Трамбле, п. Tailliez и с. Kulakauskas (1998) Estimation
of the state of the bacterial cell wall by fluorescent in situ hybridization. Заявл. Environ.
Microbiol. 64: 3059-3062.
24. Biswas, и., е. Maguin, S. D. Честно и А. приветствие (1995) A 7-base sequence pair
protects DNA from exonucleolytic деградации в Lactococcus lactis. Proc. Natl. Acad.
Sci. США 92: 2244-2248.
25. Болотин, А. с. може, к. Malarme, S. D. Честно и А. Сорокин (1999) низкий
redundancy sequencing of the entire Lactococcus lactis IL1403 геномы. AvL Int. J. Gen.
Мол. Microbiol. 76: 27-76.
26. Болотин, А., П. Wincker, С. Може, О. Jaillon, К. Malarme, J. Weiss Bach, S. D.
Честно и А. Сорокин (2001) The complete genome sequence of the lactic acid
бактерия Lactococcus lactis ssp. lactis IL1403. Genome RES. 11: 731-753.
27. Botstein, D. (1980) A theory of modular evolution for bateriophages. Ann. N. Y. Acad.
Sci. 354: 484-491.
28. Bouchard, J. D. и S. Moineau (2000) Homologous recombination between a
lactococcal bacteriophage and the chromosome of its host strain. Вирусологии 270: 65-75.
29. Буше, и., е. эмонд, э. Дион, д. Montpetit и Moineau S. (2000) Microbiological
and impacts of molecular AbiK on the lytic cycle of Lactococcus lactis phages of the 936
and P335 species. Microbiology UK 146: 445-453.
30. Коричневый, Ж. В., с. Hertwig, H. Neve, А. Гайс и тойбер м. (1989) Taxonomic
differentiation of bacteriophages of Lactococcus lactis by electron microscopy, ДНК
DNA hybridization and protein profiles. J. General Microbiol. 135: 2551-2560.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
31. Brisette, J. L., M. Russel, L. Weiner и P. Model (1990) фага shock protein, a стресс
белка of Escherichia coli. Proc. Natl. Acad. Sci. США 87: 862-866.
32. Broketa, м., А. Винс, В. Drazenovic, р. г. Сим и Mlinaric-Galinovic (2001) Non-
radioactive digoxigenin DNA labeling and immuologic detection of HSV, PCR products.
J. Clin. Virology 23: 17-23.
33. Brondsted, л. с. Ostergaard, м. Педерсен, к. молоток и ф. к. Vogensen (2001)
Analysis of the complete DNA sequence of the temperate bacteriophage TP901-1:
evolution, structure, and genome organization of lactococcal bacteriophages. Вирусологии
283: 93-109.
34. Brüssow, H. (2001a) Phages dairy of bacteria. Анну. Microbiol REV.. 55: 283-303.
35. Brüssow, ч. и ф. Desiere (2001b) Comparative фага genomics and the evolution of
Siphoviridae: insights from dairy phages. Мол. Microbiol. 39: 213-222.
36. Bruttin, А., ф. Desiere, с. Луккини, с. Фоли и е. Brussow (1997) Characterization of
the lysogeny ДНК module from the temperate Streptococcus thermophilus bacteriophage
φSfi21. Вирусологии 233: 136-148.
37. Bubendorf, Л., М. Kolmer, J. Kononen, Р. Койвисто, С. Муссы, Y. Chen, E.
Mahlamaki, с. п., ч. и Моч н. Willi (1999) гормоны failure therapy in human
prostate cancer: analysis by комплементарной ДНК and tissue microarrays. J. Natl. Cancer
Inst. 91: 1758-1764.
38. Case-Green, C. S., U. K. Мной, C. E. Pritchard и E. M. Southern (1998) Analysing
genetic information with DNA arrays. Curr. ОПИН. Chem. Biol. 2: 404-410.
39. Casjens, с. и р. Хендрикс (1988) The bacteriophages. Plenum Press, New York.
40. Casjens, S., G. Hatfull и R. W. Hendrix (1992) Evolution of dsDNA хвостатые
bacteriophage genomes. Семин. Virol. 3: 383-397.
41. Chandry, р., Б. и А. Дэвидсон Хиллер (1994a) Temporal map of the transcription
Lactococcus lactis bacteriophage sk1. Microbiology 140: 2251-2261.
42. Chandry, р. с. Moore, B. E. Davidson и А. Хиллер (1994b) Analysis of the cos области
of the Lactococcus lactis bacteriophage sk1. Гены 138: 123-126.
43. Chandry, P. S., S. C. Moore, J. D. Бойс, Б. э. Дэвидсон и A. J. Hillier (1997)
Analysis of the последовательности ДНК, экспрессии генов, origin of replication and molecular
structure of the Lactococcus lactis lytic bacteriophage sk1. Мол. Microbiol. 26: 49-64.
44. Chee, M., R. Yang, Е. Hubbell, А. Berno, X. C. Huang, D. Stern, J. Winkler, D. J.
Локхарт, Morris M. S. S. p. a. и Фодор (1996) доступ к любимым геномной information with
высокая плотность массивов. Science 274: 610-614.
45. Ченг, с., с. Fockler, W. M. Barnes и R. Higuchi (1994) Effective amplification of
long targets from cloned and inserts геномной ДНК человека. Proc. Natl. Acad. Sci. США
91: 5695-5699.
46. Шевалье, Б. с. и Б. л. Стоддард (2001) Homing endonucleases: structural and
functional insight into the катализаторы of Интрон/intein mobility. Nucl. Acids RES. 29: 3757-
3774.
47. Шопен, м. с., А. Шопен, Руо А. н. и Galleron (1989) and Insertion
amplification of foreign genes in the Lactococcus lactis subsp. lactis хромосомы. Заявл.
Environ. Microbiol. 55: 1769-1774.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
48. Шопен, А. (1993) Organization and regulation of genes for amino acid biosynthesis в
lactic acid bacteria. FEMS Microbiol. ОТКР. 12: 21-38.
49. Шопен, А. А. Болотин, А. Сорокин, с. д. Честно и м.-в. Шопен (2001) Analysis of
six prophages в Lactococcus lactis IL1403: different genetic structure of temperate and
вирулентного фага populations. Nucl. Acids RES. 25: 644-651.
50. Chung, D. K., J. H. Kim и C. A. Batt (1991) Cloning and nucleotide sequence of the
майор capsid protein from Lactococcus lactis ssp. cremoris bacteriophage F4-1. Гены
101: 121-125.
51. Cluzel, P. J., A. Шопен, с. д. Честно и Шопен м. к. (1991) фага абортивных infection
mechanism from Lactococcus lactis subsp. lactis, expression of which is mediated by an
Iso-IS S1 элемент. Заявл. Environ. Microbiol. 57: 3547-3551.
52. Coakley, M., G. F. Fitzgerald и R. P. Ross (1997) Application and evaluation of the
фаговая resistance and bacteriocin-encoding плазмиды pMRC01 for the improvement of
dairy starter cultures. Заявл. Environ. Microbiol. 63: 1434-1440.
53. Cocconcelli, п. с., д. порро, с. Galandini и л. Senini (1995) Development of a RAPD
protocol for typing of strains of lactic acid bacteria and enterococci. Lett. Заявл.
Microbiol. 21: 376-379.
54. Коффи, А., м. Coakley, А. Макгарри, G. F. Fitzgerald и R. P. Ross (1998) Increasing
фаговая resistance of cheese starters - a case study using Lactococcus lactis DPC4268.
Lett. Заявл. Microbiol. 26: 51-55.
55. Коффи, А., д. Стоукс, G. F. Fitzgerald и R. P. Ross (2001) Traditional and molecular
approaches to improving bacteriophage resistance of Cheddar and Mozzarella cheese
starters. Irish J. Agricul. Food RES. 40: 239-270.
56. Коган, Т. М., М. Барбоза, Э. Beuvier, В. Бианки Salvadori, P. S. Cocconcelli, И.
Фернандеш, Ж. Гомес, Р. Гомес, Г. Kalantzopoulos, А. Ледда, М. Медина, М. С. Rea
и э. Родригес (1997) Characterization of the lactic acid bacteria in dairy artisanal
products. J. Dairy RES. 64: 409-421.
57. Corpet, F. (1988) Multiple sequence alignment with hierarchical clustering. Nucl. Acids
RES. 22: 10881-10890.
58. Corroler, д., н. Desmasures и м. Gueguen (1999) Correlation between полимеразы
chain reaction analysis of the histidine biosynthesis operon, randomly amplified
polymorphic DNA analysis and phenotypic characterization of dairy Lactococcus
универсальные подходят. Заявл. Microbiol. Biotechnol. 51: 91-99.
59. Crow, V. L., T. Cool Bear, р. к. Гопал, ф. г. Martley, л. л. и х. Маккей Riepe (1995a)
The role of autolysis of lactic acid bacteria in the ripening of cheese. Int. J. Dairy 5: 855-
875.
60. Ворона, В. л., р. к. Гопал и А. я. Вика (1995b) Cell surface of differences
lactococcal strains. Int. J. Dairy 5: 45-68.
61. Ворона, л. В., ф. г. Martley, T. Cool Bear и S. J. round Hill (1995c) The influence of
фага-assisted лизиса of Lactococcus lactis subsp. lactis Ml8 on cheddar cheese ripening.
Int. J. Dairy 5: 451-472.
62. Crutz-Le Coq, А. м., Б. Cesselin, J. Комиссар и J. Anba (2002) Sequence analysis
of the lactococcal bacteriophage bIL170: insights into structural proteins and HNH
endonucleases в dairy phages. Microbiology 148: 985-1001.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
63. Daly, C. (1983) The use of mesophilic cultures in the dairy industry. А. в. Leeuwenhook
Int. J. Gen. M. 49: 297-312.
64. Daly, C. G. F. Fitzgerald и R. Davis (1996) Biotechnology of lactic acid bacteria with
special reference to bacteriophage resistance. А. в. Левенгук Int. J. Gen. M. 70: 99-
110.
65. Daly, C. G. F. Fitzgerald, L. O. о'Коннор и р. Дэвис (1998) Technological and health
benefits of dairy starter cultures. Int J Dairy 8: 195-205.
66. Давидсон, А. р. и м. Gold (1992) Мутационной abolishing the activity of endonuclease
bacteriophage lambda terminase lie in two distinct regions of the гены A, one of which
may encode a "Leucine Zipper" ДНК-связывающий домен. Вирусологии 189: 21-30.
67. Дэвидсон, Б. э. и А. Дж. Хиллер (1995) Developing new for starters ферментированные молоко
products. Aust. J. Dairy Технол. 50: 6-9.
68. Дэвис, ф. л., х. м. и м. Андервуд Дж Gasson (1981) The value of плазмиды profiles
for strain identification в lactic streptococci and the relationship between Streptococcus
lactis 712, ML3 и с2. J. Appl. Bacteriol. 51: 325-337.
69. Дэвис, р., д. Vanderlelie, А. Mercenier, C. Daly и г. ф. Фицджеральда (1993) Scr FI
restriction-modification system of Lactococcus lactis subsp. cremoris UC503 - cloning
and characterization of two Scr FI methylase genes. Заявл. Environ. Microbiol. 59: 777-
785.
70. де Ман, Дж. к., Рогоза м. и M. E. Sharpe (1960) A medium for the выращивание грибов of
lactobacilli. J. Appl. Bacteriol. 23: 130-135.
71. ru Risi, J. L., V. R. Iyer и P. O. Brown (1997) Exploring the metabolic and genetic
control of gene expression patterns with a комплементарной ДНК microrarray. Science
270: 680-686.
72. de Ruyter, п. г., о. п. Кейперсом, W. C. Meijer и de Vos W. M. (1997) Food-grade
controlled лизиса of Lactococcus lactis for accelerated cheese ripening. Nat. Biotechnol.
15: 976-979.
73. ru Saizieu, A., & Certa, J. Warrington, C. Gray, W. Keck и J. Mous (1998) Bacterial
transcript imaging by hybridization of total RNA to олигонуклеотид массивов. Nat.
Biotechnol. 16: 45-48.
74. де Вос, в. м., п. Tesino, р. Ван краненбург, е. Luesink, о. п. Кейперсом, J. van de Roost,
М. Kleerebezem и J. Hugenholtz (1998) Making more of milk sugar by engineering
lactic acid acteria. Int. J. Dairy 8: 227-233.
75. Delorme, C., J. J. ГОДО, S. D. Честно и п. Renault (1994) Mosaic structure of large
regions of the Lactococcus lactis subsp. cremoris хромосомы. Microbiology UK 140:
3053-3060.
76. Deng, в., В. Burland, г. р. Планкетт, А. Boutin, г. ф. Мэйхью, р. Лисс, н. Перна т. д.
J. Rose, B. МАУ, S. Zhou, D. C. Schwartz, J. D. Fetherston, Л. Е. Lindler, Р. Р.
Брубейкер, Г. В. Плано, S. C. Straley, К. А. МакДоноу, М. Л. Nilles, И. С. Мэтсон Р. Ф.
Blattner и R. D. Perry (2002) Genome sequence of Yersinia pestis KIM. J. Bacteriol.
184: 4601-4611.
77. Дэн, Y. M., M. L. Harvey, Q. C. Liu и N. W. Dunn (1997) A novel плазмиды-encoded
фаговая абортивных system from infection Lactococcus lactis биовар. diacetylactis. FEMS
Microbiol. Lett. 146: 149-154.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
78. Deng, Y. M. C. Q. N. W. Liu и Dunn (1999) Genetic and functional organization
analysis of a novel фага абортивных infection системы, AbiL, from Lactococcus lactis. J.
Biotechnol. 67: 135-149.
79. Descheemaeker, р., Б. Банк, А. м. Ledeboer, т. Verrips и к. Kersters (1994)
Comparison of the Lactococcus lactis дифференциальной средой (DCL) и SDS-page of
whole-cell proteins for the identification of lactococci to subspecies уровня. Syst. Заявл.
Microbiol. 17: 459-466.
80. Desiere, ф., с. Mahanivong, A. J. Hillier, P. S. Chandry, Б. э. Дэвидсон и х. Brüssow
(2001) Comparative genomics of lactococcal phages: insight from the complete genome
sequence of Lactococcus lactis фага BK5-T. Вирусологии 283: 240-252.
81. Динсмор, P. K. и T. R. Klaenhammer (1994) Phenotypic consequences of altering the
copy number of Авия, a gene responsible for aborting bacteriophage infections in
Lactococcus lactis. Заявл. Environ. Microbiol. 60: 1129-1136.
82. Динсмор, P. K. и T. R. Klaenhammer (1997) Molecular characterization of a геномной
регион в a Lactococcus bacteriophage that is involved in its sensitivity to the фага
defense mechanism Авия. J. Bacteriol. 179: 2949-2957.
83. Джорджевич, G. M. и T. R. Klaenhammer (1997) Bacteriophage-triggered обороны
системы - фага adaptation and design improvements. Заявл. Environ. Microbiol. 63:
4370-4376.
84. Джорджевич, G. M., D. J. о'Салливан, с. А. Уолкер, м. А. Conkling и т. р.
Klaenhammer (1997) A triggered-suicide system designed as a defense against
bacteriophages. J. Bacteriol. 179: 6741-6748.
85. Догерти, Б. А., к. Хилл, Дж. Ф. Weidman, D. R. Richardson, J. C. Venter и р. п. Росс
(1998) Sequence and analysis of the 60 КБ conjugative, bacteriocin-producing плазмиды
pMRC01 from Lactococcus lactis DPC3147. Мол. Microbiol. 29: 1029-1038.
86. Дагган, D. J., M. Биттнер, Y. Chen, P. J. M. Meltzer и Трент (1999) экспрессия
профилирование using cDNA microarrays. Suppl. Nat. Genet. 21: 10-14.
87. Дюплесси, м. и с. Moineau (2001) Identification of a genetic determinant responsible
for host specificity in Streptococcus thermophilus bacteriophages. Мол. Microbiol. 41:
325-336.
88. Дурмаз, э., л. д. Хиггинс и Klaenhammer T. R. (1992) Molecular characterization of
a 2nd абортивных фага resistance gene в present Lactococcus lactis subsp. lactis ME2. J.
Bacteriol. 174: 7463-7469.
89. Дурмаз, э. и Klaenhammer T. R. (1995) A starter culture rotation strategy
Каспия paired restriction/modification and абортивных infection bacteriophage
Оборона in a single Lactococcus lactis штамм. Заявл. Environ. Microbiol. 61: 1266-1273.
90. Дурмаз, э. и Klaenhammer T. R. (2000) Genetic analysis of сцепленные of regions
Lactococcus lactis acquired by recombinant lytic phages. Заявл. Environ. Microbiol. 66:
895-903.
91. Edgell, д. р. и д. А. Shub (2001) Related самонаведения endonucleases I- Bmo I and I- ТРВ I
use different strategies to cleave homologous recognition sites. Proc. Nat. Acad. Sci. США
98: 7898-7903.
92. el Karoui, M., D. Честно и А. приветствие (1998) Identification of the lactococcal
exonuclease/рекомбинантному and its modulation by the предполагаемых чи sequence. Proc. Nat.
Acad. Sci. США 95: 626-631.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
93. Эмонд, э., Б. Хеллер, и. Буше, п. Ванденберг, э. Vedamuthu, Kondo J. и S.
Moineau (1997) Phenotypic and genetic characterization of the bacteriophage абортивных
infection mechanism abiK from Lactococcus lactis. Заявл. Environ. Microbiol. 63: 1274-
1283.
94. Эмонд, э., э. Дион, с. А. Уолкер, э. р. Vedamuthu, J. K. Kondo и Moineau S. (1998)
AbiQ, абортивных infection mechanism from Lactococcus lactis. Заявл. Environ.
Microbiol. 64: 4748-4756.
95. Энгельберг-кулка, х. и г. Глазер (1999) Addiction modules and programmed cell
death and Anti Death in bacterial cultures. Анну. Microbiol REV.. 53: 43-70.
96. Engelke, г., З. Гутовский Eckel, Баранов м. человек и к. д. Entian (1992) Biosynthesis
of the lantibiotic низин - геномной organization and membrane localization of the NisB
белка. Заявл. Environ. Microbiol. 58: 3730-3743.
97. Эпштейн, с. Б. и Butow R. A. (2000) Microarray technology - enhanced versatility,
постоянный challenge. Curr. ОПИН. Biotechnol. 11: 36-41.
98. Erlandson, к. и с. А. Батт (1997) Strain-specific differentiation of lactococci в смешанных
starter culture using populations randomly amplified polymorphic DNA derived probes.
Заявл. Environ. Microbiol. 63: 2702-2707.
99. Эспозито, д., в. п. Fitzmaurice, Benjamin R. C., S. D. Goodman, A. S. и J. Waldman
J. Scocca (1996) The complete nucleotide sequence of bacteriophage HP1 ДНК. Nucl.
Acids RES. 24: 2360-2368.
100. Farber J. M. (1996) An introduction to the hows and whys of molecular typing. J. Food
Protection 59: 1091-1101.
101. Ferea, T. L. и P. O. Brown (1999) Observing the living геномы. Curr. ОПИН. Genet.
Dev. 9: 715-722.
102. Ferretti, J. J., W. M. McShan, Д. Adjic, Д. Савич, Г. Г. Савич, К. Лион, C. Примо, С. С.
Sezate, А. Н. Surorov, С. Кентон, Lai H., S. Lin, Y. Qian, H. G. Jia, Ф. Э. Нахар, Кв. Рен,
H. Zhu, л. песня, J. White, X. Yuan, Клифтон с. в., Б. А. Роу и р. э. Маклафлин
(2001) Complete genome sequence of an M1 strain of Streptococcus pyogenes. Proc.
Natl. Acad. Sci. США 98: 4658-4663.
103. Флейшман, R. D., M. D. Adams, О. Уайт, Р. А. Клейтон, Э. Ф. Kirkness, А. Р.
Kerlavage, C. J. Bult, Tomb и. ф., Б. А. и J. Dougherty м. Меррик (1995) Whole
геномы random sequencing of Haemophilus influenzae Rd. Science 269: 496-512.
104. Фоли, с., с. Луккини, м. с. Zwahlen и H. Brussow (1998) A short noncoding viral
ДНК элемент showing characteristics of a replication origin confers bacteriophage
resistance to Streptococcus thermophilus. Вирусологии 250: 377-387.
105. Фоли, с., А. Bruttin и H. Brussow (2000) Widespread distribution of a group I Интрон
and its three deletion in the производные лизина гены of Streptococcus thermophilus
bacteriophages. J. Virology 74: 611-618.
106. Ферде, А. и г. ф. Фицджеральда (ICRP, 1999a) Analysis of exopolysaccharide (EPS) производства
mediated by the bacteriophage адсорбции blocking плазмиды pCI658 isolated from
Lactococcus lactis ssp. cremoris HO2. Int. J. Dairy 9: 465-472.
107. Ферде, А. и г. ф. Фицджеральда (1999b) Bacteriophage defence systems в lactic acid
bacteria. А. в. Левенгук Int. J. Gen. Mol. Microbiol. 76: 89-113.
108. Фридрих, J. E. и T. E. Acree (1998) Gas chromatography olfactometry (GC/O) of
dairy products. Int. J. Dairy 8: 235-241.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
109. Фукс, Б. м., г. Wallner, в. Beisker, и. Schwippl, в. Людвиг и Amann R. (1998)
Flow cytometric analysis of the in situ accessibility of Escherichia coli 16S rRNA for
fluorescently labeled олигонуклеотидных зондов. Заявл. Environ. Microbiol. 64: 4973-4982.
110. Гвардия, с. м. Бабин, р. Гая, м. Нуньес, м. Медина (1999) PCR amplification of the
гены acmA differentiates Lactococcus lactis subsp. lactis и L. lactis subsp. cremoris.
Заявл. Environ. Microbiol. 65: 5151-5153.
111. Гарви, п., г. ф. с. Фицджеральд и Хилл (1995a) Cloning and DNA sequence analysis of
two абортивных infection фага resistance from the determinants плазмиды lactococcal
pNP40. Заявл. Environ. Microbiol. 61: 4321-4328.
112. Гарви, P., D. van Sinderen, д. п. Туми, C. Hill и г. ф. Фицджеральда (1995b)
Molecular genetics of bacteriophage and natural фага defense systems in the genus
Lactococcus. Int. J. Dairy 5: 905-947.
113. Гарви, п., с. Хилл и г. ф. Фицджеральда (1996) The lactococcal плазмиды pNP40 a кодирует
3й bacteriophage resistance mechanism, one which affects ДНК фага проникновения.
Заявл. Environ. Microbiol. 62: 676-679.
114. Гарви, п., А. промывка IT OFF, C. Hill и г. ф. Фицджеральда (1997) Identification of a RecA
гомолог (RecALp) on the conjugative lactococcal фага resistance плазмиды pNP40 -
evidence of a role for chromosomally encoded RecAL в абортивных infection. Заявл.
Environ. Microbiol. 63: 1244-1251.
115. Gasson, M. J. (1983) Genetic transfer systems в lactic acid bacteria. А. в. Левенгук
Int. J. Gen. M. 49: 275-282.
116. Gasson, M. J., S. Swindell, Maeda S. и H. M. Dodd (1992) Molecular rearrangement
of лактозы ДНК плазмиды associated with high-frequency transfer and cell aggregation в
Lactococcus lactis 712. Мол. Microbiol. 6: 3213-3223.
117. Gasson, M. J. (1993) Progress and potential in the biotechnology of lactic acid bacteria.
FEMS Microbiol. ОТКР. 12: 3-20.
118. Gasson, M. J., J. J. ГОДО, C. J. Pillidge, T. J. Eaton, к. жюри и с. А. Ширман (1995)
Characterization and exploitation of conjugation в Lactococcus lactis. Int. J. Dairy 5:
757-762.
119. Gasson, M. J. (1996a) Lytic системы lactic acid bacteria and their bacteriophages. А. в.
Левенгук Int. J. Gen. Microbiol. 70: 147-159.
120. Gasson, м. и., Бенсон к., с. Swindell и H. Griffin (1996b) Metabolic engineering of
the Lactococcus lactis диацетил pathway. Lait 76: 33-40.
121. Геринга, к., А. Charbit, E. Brissaud и м. Hofnung (1987) Bacteriophage receptor site
on the Escherichia coli K-12 LamB белка. J. Bacteriol. 169: 2103-2106.
122. Geiss, G. K., R. E. Bumgarner, м. с., м. Б. Agy, A. B. van't Wout, E.
Молоток Марк, В. S. Carter, D. Upschurch, Mullins J. I. и M. G. Кот (2000) Large
scale monitoring of host cell gene expression during HIV-1 infection using cDNA
microarrays. Вирусологии 266: 8-16.
123. Геллер, Б. л., р. г. Айви, J. E. Trempy и Б. hettinger Смит (1993) Cloning of a
сцепленные гены required for фага infection of Lactococcus lactis subsp. lactis с2. J.
Bacteriol. 175: 5510-5519.
124. Глазер, Р., Л. Frangeul, С. Книга Rieser, С. Rusniok, А. Изменить, Ф. Бакеро, Р. Berche, H.
Bloecker, р. Брандт, т. Чакраборти, А. Charbit, ф. Chetouani, е. Кув, А. де даруваре,
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
P. Dehoux, Е. Domann, Г. Dominguez-Берналь, Э. Duchaud, Л. Дюрант, О. Dussurget, К.
Entian, H. Fsihi, Ф. Портильо, Р. Гарридо, Л. Готье, В. Гебель, Н. Гомес-Лопес, T.
Роща, J. Чна, Д. Джексон, Л. Джонс, & Kaerst, J. Крэфт, М. Кун, Ф. Искусства, Г. Kurapkat,
E. Madueno, А. Maitournam, Vicente J. M., Ng E., H. Nedjari, Г. Nordsiek, Новелла С.,
B. de Pablos, J. C. Перес-Диас, р. Перселл, Б. Remmel, м. Роуз, т. Schlueter, Simoes н.,
А. Tierrez, J. A. Vazquez-Boland, H. Voss, J. Wehland и п. коссар (2001)
Comparative genomics of Listeria species. Science 294: 849-852.
125. Глазго, A. C. (1989) Mobile DNA, р. 637-659, в Berg D. E. и M. M. Howe, eds.
Мобильные ДНК. American Society for Microbiology Press, Washington, D. C.
126. Горбун, F. O. (1998) филогения и in situ -определение прокариот в
limnischen и морских Экосистем. Докторская Диссертация, Технический Университет Мюнхена,
Кафедра микробиологии, Мюнхен.
127. Горбун, ф. о., Б. м. Фокс и р. Amann (1999) Bacterioplankton compositions в
lakes and oceans: a first comparison based on fluorescence in situ hybridization. Заявл.
Environ. Microbiol. 65: 3721-3726.
128. Golub, T. R., D. K. Слоним, Р. Тамайо, К. Huard, М. Gaasenbeck, J. P. Mesirov, H.
Коллер, м. л. Loh, J. R. Downing и M. A. Caligiuri (1999) Molecular classification of
cancer: class discovery class and prediction by gene expression мониторинга. Science 286:
531-537.
129. Goodrich-Blair, х. и д. А. Shub (1994) The ДНК полимеразы of genes serveral HMU
bacteriophages have similar group I интронов with highly divergent open reading frames.
Nucl. Acids RES. 22: 3715-3721.
130. Goodrich-Blair, х. и д. А. Shub (1996) Beyond самонаведения: competition between Интрон
endonucleases confers a selective advantage on flanking genetic markers. Cell 84: 211-
221.
131. Хаггард-Ljungquist, е., с. Halling и R. Calendar (1992) DNA sequence of the tail
волоконно-genes of bacteriophage Р2: evidence for horizontal transfer of fiber tail genes
among несвязанные bacteriophages. J. Bacteriol. 174: 1462-1477.
132. Harrington, C. A., C. Rosenow и Ж.-ретифе (2000) мониторинг экспрессии генов using
DNA microarrays. Curr. ОПИН. Microbiol 3: 285-291.
133. Hattman, с. (1970) метилирования ДНК of T even bacteriphages and their non-
glucosylated mutants: its role in P1-directed restriction. Вирусологии 42: 359-367.
134. Хейворд, R. E., J. L. De ризи, с. Alfadhli, D. C. Kaslow, P. O. п. Браун и к. Rathod
(2000) Shotgun DNA microarrays and stage-specific gene expression in Plasmodium
falciparum малярии. Мол. Microbiol. 35: 6-14.
135. Кучи, H. A. и R. C. Lawrence (1976) The selection of стартер strains for cheesemaking.
N. Z. J. Dairy Sci. Technol. 2: 16-20.
136. Хеллер, к. Ю., А. Гайс и H. Neve (1995) Behaviour of genetically modified
Micro орган в Sims йогурт. Syst. Заявл. Microbiol. 18: 504-509.
137. Хендрикс, R. W., M. C. M. Smith, R. N. Burns, M. E. Ford и г. ф. Hatfull (1999)
Evolutionary relationships among различные bateriophages and prophages: all the world's a
фага. Proc. Natl. Acad. Sci. США 96: 2192-2197.
138. Hertwig, с., в. bockel мужчина и тойбер м. (1997) Purification and characterization of
the lytic induced activity by the prolate-headed bacteriophage P001 в Lactococcus
lactis. J. Appl. Microbiol. 82: 233-239.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
139. Хилл, с., л. А. Миллер и Klaenhammer T. R. (1990) Cloning, expression, and sequence
детерминации of a bacteriophage фрагмент encoding bacteriophage в resistance
Lactococcus lactis. J. Bacteriol. 172: 6419-6426.
140. Хилл, с., л. А. Миллер и Klaenhammer T. R. (1991) In vivo genetic exchange of a
functional for domain between lactococcal плазмиды pTR2030 and a virus. J. Bacteriol.
173: 4363-4370.
141. Hill, C. (1993) and Bacteriophage bacteriophage в resistance lactic acid bacteria. FEMS
Microbiol. ОТКР. 12: 87-108.
142. Хо, Y., Kim S. J. и р. Б. варинг (1997) A protein encoded by a group I Интрон в
Aspergillus nidulans directly передач RNA splicing and is a DNA endonuclease. Proc.
Natl. Acad. Sci. США 94: 8994-8999.
143. Хольцапфель, W. H. и U. шиллингер (1993) The Genus Leuconostoc, р. 1508-1534, А. в
Ballows, H. Trüper, м. Дворкин, в. Хардер, и к. мясорубки, eds. The Prokaryotes.
Springer Verlag, New York.
144. Huang, D. C., M. Novel г. и Novel (1991) A transposon-like element on the лактозы
плазмиды of Lactococcus lactis subsp. lactis Z270. FEMS Microbiol. Lett. 77: 101-106.
145. Hugenholtz, J. (1993) Цитрат metabolism в lactic acid bacteria. FEMS Microbiol. REV.
12: 165-178.
146. Hugenholtz, р., Б. м. и н. Гебель р. Pace (1998) Impact of culture-independent
studies on the emerging phylogenetic view of bacterial diversity. J. Bacteriol. 180: 4765-
4774.
147. Хаггинс, A. R. (1984) Progress in dairy starter culture technology. Food Technol. 38: 41-
50.
148. Халл, р. р. (1977) Methods for monitoring bacteriophage в cheese factories. Aust. J.
Dairy Технол. 32: 63-64.
149. Голод, W. (1986) бактериофаги в молокоперерабатывающей промышленности. Немецкие
Молокозавод-газета DMZ 51/52: 1762-1769.
150. Husson-Kao, C. J. Mengaud, J. C. Gripon, л. Benbadis и м. п. Chapot-Шартье
(1999) The autolysis of Streptococcus thermophilus DN-001065 is triggered by several
food-grade environmental сигнала. Int. J. Dairy 9: 715-723.
151. Husson-Kao, C. J. Mengaud, Б. Cesselin, D. van Sinderen, л. Benbadis и м. п.
Chapot-Шартье (2000) The Streptococcus thermophilus autolytic phenotype results from
дырявый prophage. Заявл. Environ. Microbiol. 66: 558-565.
152. IRP, U. (1998) для исследования молекулярно-генетических Конституции выбранных
Молочнокислых бактерий-изолятов от мрачной культур для производства ферментированного
Молочные продукты. Дипломная Работа, Бременский Университет, Биотехнологии И Молекулярной
Генетика, Бремен.
153. Iwahana, H., M. Fujimura, Y. Takahashi, T. Iwabuchi, к. Есимото и Итакура м.
(1996) Multiple fluorescence-based PCR-SSCP analysis using fluorescent internal
labeling of PCR products. Напряжение 21: 510-519.
154. Джарвис, А. В., Г. Ф. Фицджеральд, М. Мата, А. Mercenier, H. Neve, I. B. Powell, C. Ронда,
М. Saxelin и тойбер м. (1991) Species and type phages of lactococcal bacteriophages.
Интер вирусологии 32: 2-9.
6. Литература
_____________________________________________________________________________________________________________________________________________________________________________________
155. Джарвис, А. в., м. в. лубберса, N. R. Waterfield, L. J. Collins и к. м. Polzin (1995)
Sequencing and analysis of the genome of lactococcal фага с2. Int. J. Dairy 5: 963-976.
156. Josephsen,