Официальные источники
Если вы проживаете не в России, обратитесь к аналогичным сайтам вашей страны.
Мойте руки, берегите близких, по возможности оставайтесь дома и работайте удалённо.
Читать публикации про: коронавирус | удалённую работу

Ши Чжэнли в своей лаборатории в Уханьском институте вирусологии
Но прежде чем разводить тут конспирологию, давайте сначала окунёмся в биологию.
Биология
Итак, начнём от печки. Что за фуриновый сайт, что за RBM, что вообще за шиповидный белок? На самом деле, если продраться сквозь дебри терминологии, то концептуально всё просто. Вот, например, шиповидный белок — это та самая торчащая из вирусной частицы штука (S protein), за которую эти вирусы и “короновали”:

Именно с помощью этих белков вирион цепляется за рецептор клетки-жертвы (ACE2 в нашем случае), чтобы затем проникнуть внутрь. Поэтому можно сказать, что это самая важная часть вируса, ведь именно она определяет каких животных тот может поражать, а каких нет — ACE2 рецепторы у разных видов немного структурно отличаются. При этом из всего огромного по вирусным меркам 30-килобазного генома, ген этого белка составляет лишь 12–13%, то есть около 1300 аминокислот. Вот так структурирован шиповидный белок у CoV2 и его близких родственников:
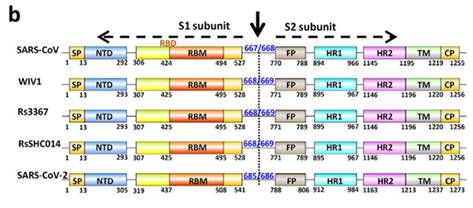
Как видно из рисунка выше, S белок состоит из двух субъединиц: S1 и S2. Именно S1 взаимодействует с рецептором ACE2, и то место, которым он это делает, называется Receptor Binding Domain (RBD), а область непосредственного контакта, святая святых, называется Receptor Binding Motif (RBM). Вот красивая иллюстрация из не менее красивой работы:
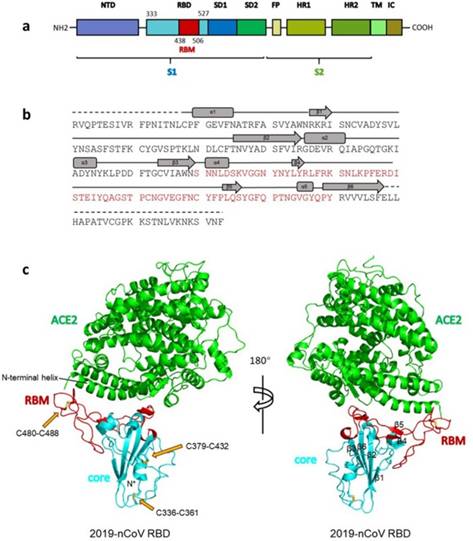
Общая структура RBD CoV2, связанного с ACE2.
(а) Общая топология мономера шиповидного белка 2019-nCov. NTD, N-терминальный домен. RBD, рецептор-связывающий домен. RBM, рецептор-связывающий мотив. SD1, поддомен 1. SD2, поддомен 2. FP, пептид слияния. HR1, гептадный повтор 1. HR2, гептадный повтор 2. TM, трансмембранная область. IC, внутриклеточный домен.
(б) Последовательность и вторичные структуры RBD 2019-nCov. RBM окрашен в красный цвет.
(с) Общая структура RBD 2019-nCov, связанного с ACE2. ACE2 окрашен в зеленый цвет. Ядро RBD 2019-nCoV окрашено в голубой цвет, а RBM — в красный. Дисульфидные связи в RBD 2019-nCov показаны в виде линии и обозначены желтыми стрелками. N-концевая спираль ACE2, ответственная за связывание, помечена.
Исходный текст
Overall structure of 2019-nCoV RBD bound with ACE2.
(a) Overall topology of 2019-nCoV spike monomer. NTD, N-terminal domain. RBD, receptor-binding domain. RBM, receptor-binding motif. SD1, subdomain 1. SD2, subdomain 2. FP, fusion peptide. HR1, heptad repeat 1. HR2, heptad repeat 2. TM, transmembrane region. IC, intracellular domain.
(b) Sequence and secondary structures of 2019-nCoV RBD. The RBM is colored red.
(с) Overall structure of 2019-nCoV RBD bound with ACE2. ACE2 is colored green. 2019-nCoV RBD core is colored cyan and RBM is colored red. Disulfide bonds in the 2019-nCoV RBD are shown as stick and indicated by yellow arrows. The N-terminal helix of ACE2 responsible for binding is labeled.
Так вот. Когда геном CoV2 только расшифровали, то сначала не было известно каких-то прям близкородственных ему штаммов. Но уже 23 января 2020 года Ши Чжэнли выпустила работу, в которой объявила, что CoV2 на 96% совпадает со штаммом RaTG13, который её лаборатория в 2013 году выделила из Юньнаньских летучих мышей. Правда, вне её лаборатории до января 2020 года об этом штамме не было известно никому.
Было сразу понятно, что RaTG13 — мальчик особенный. Взгляните на график:
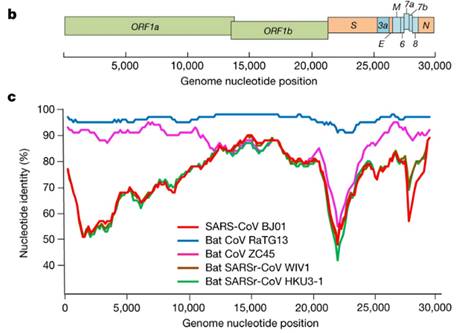
Это график схожести CoV2 с другими известными штаммами. Чем выше кривая, тем выше процент совпадения нуклеотидов. Как видим, в районе гена того самого шиповидного белка (S) только RaTG13 более-менее близок к CoV2, а все остальные штаммы в этом месте уходят в пике — как штаммы вирусов из других летучих мышей, так и первый SARS-CoV (красная кривая). Но тут пока нет ничего подозрительного — мало ли ещё неизвестных науке штаммов таят в себе неизведанные Юньнаньские пещеры? Ну да, не очень понятно как именно вирус оттуда добрался до Уханя, но чего не бывает.
Панголины
Далее на сцене появляются панголины: в феврале другая группа китайских учёных обнаружила в своих закромах штамм панголиньего коронавируса, который, хоть в целом и хуже чем RaTG13 был похож на CoV2 (на 90%), в том самом RBM шиповидного белка был почти идентичен — отличался лишь на 1 аминокислоту (см. два верхних сиквенса, точки означают совпадение с первым сиквенсом):

При этом в первой четверти S белка панголиний штамм на CoV2 не похож, а последовательность после RBM у всех троих штаммов (CoV2, Pangolin, RaTG13) более-менее совпадает. Но вот сам RBM у RaTG13 весьма отличается от CoV2, что видно по крутому провалу зелёного графика RaTG13 по сравнению с красным графиком CoV2 в районе RBM (вертикальная розовая полоса) на следующем графике:
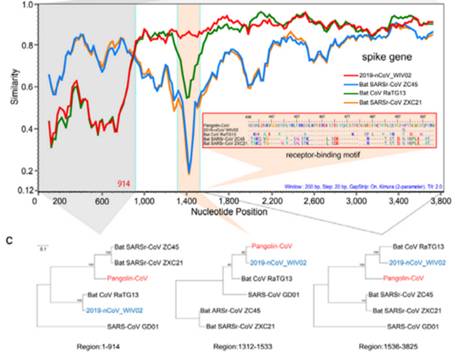
Эту разницу подтверждает и филогенетический анализ этих трёх областей, выделенных на графике выше — по RBM штамм панголина ближе к CoV2, чем RaTG13, а вот слева и справа от RBM к CoV2 ближе именно RaTG13. То есть налицо явная рекомбинация, о чем упоминают и сами авторы, и некоторые другие работы.
Кстати, откуда взялись у исследователей эти самые панголины? А вот отсюда:

Их конфисковала у контрабандистов китайская таможня и передала в реабилитационный центр в Гуандуне, где они скончались при выраженной коронавирусной симптоматике. Это, конечно, не могло не заинтересовать местных вирусологов, которые и выделили у них различный биоматериал:
Панголины, использованные в исследовании, были конфискованы таможней и Департаментом лесного хозяйства провинции Гуандун в марте-декабре 2019 года. В их число входят четыре китайских панголина (Manis pentadactyla) и 25 малайских панголинов (Manis javanica). Эти животные были отправлены в центр спасения диких животных; в основном они были неактивны и задыхались, и в конечном итоге умерли, несмотря на обширные усилия по их спасению. Образцы тканей были взяты из легких, лимфатических узлов, печени, селезенки, мышц, почек и других тканей у только что умерших панголинов, для гистопатологических и вирусологических исследований.
Исходный текст
Pangolins used in the study were confiscated by Customs and Department of Forestry of Guangdong Province in March-December 2019. They include four Chinese pangolins (Manis pentadactyla) and 25 Malayan pangolins (Manis javanica). These animals were sent to the wildlife rescue center, and were mostly inactive and sobbing, and eventually died in custody despite exhausting rescue efforts. Tissue samples were taken from the lung, lymph nodes, liver, spleen, muscle, kidney, and other tissues from pangolins that had just died for histopathological and virological examinations.
Кстати, и не только местных, потому что другие китайские исследователи (гонконгские в данном случае) тоже получали образцы конфискованных панголинов и в феврале 2020 тоже выпустили схожую работу, отметив явные признаки рекомбинации в шиповидном белке CoV2:
Мы получили образцы замороженных тканей (легких, кишечника, крови), которые были взяты у 18 малайских панголинов (Manis javanica) в течение августа 2017 года — января 2018 года. Эти панголины были получены в ходе операций по борьбе с контрабандой таможней Гуанси. Поразительно, что высокопроизводительное секвенирование их РНК выявило присутствие коронавирусов в шести (два легких, два кишечника, одна смесь легких и кишечника, одна кровь) из 43 образцов.
…
Более заметным, однако, было наблюдение предполагаемых сигналов рекомбинации между коронавирусами панголинов, коронавирусами летучих мышей RaTG13 и 2019-CoV2 человека (Рис. 1c, d). В частности, 2019-CoV2 демонстрирует очень высокое сходство последовательностей с коронавирусами панголина из провинции Гуандун в рецептор-связывающем домене (RBD; сходство аминокислот 97,4%; обозначено красной стрелкой на рис. 1c и рис. 2a), хотя в оставшейся части вирусного генома 2019-CoV2 ближе всего к RaTG13. В то же время RaTG13 и 2019-CoV2 имеют только 89,2% сходства аминокислот в RBD. Коронавирусы панголинов из Гуандуна и 2019-CoV2 содержат идентичные аминокислоты в пяти критических остатках RBD, тогда как RaTG13 имеет только одну общую аминокислоту с 2019-CoV2 из этих пяти (остаток 442, нумерация SARS-CoV человека).
Исходный текст
We received frozen tissue (lungs, intestine, blood) samples that were collected from 18 Malayan pangolins (Manis javanica) during August 2017-January 2018. These pangolins were obtained during the anti-smuggling operations by Guangxi Customs. Strikingly, high-throughput sequencing of their RNA revealed the presence of coronaviruses in six (two lung, two intestine, one lung-intestine mix, one blood) of 43 samples. With the sequence read data, and by filling gaps with amplicon sequencing, we were able to obtain six full or nearly full genome sequences — denoted GX/P1E, GX/P2V, GX/P3B, GX/P4L, GX/P5E and GX/P5L — that fall into the 2019-CoV2 lineage (within the genus Betacoronavirus) in a phylogenetic analysis (Figure 1a).
…
More notable, however, was the observation of putative recombination signals between the pangolins coronaviruses, bat coronaviruses RaTG13, and human 2019-CoV2 (Figure 1c, d). In particular, 2019-CoV2 exhibits very high sequence similarity to the Guangdong pangolin coronaviruses in the receptor-binding domain (RBD; 97.4% amino acid similarity; indicated by red arrow in Figure 1c and Figure 2a), even though it is most closely related to bat coronavirus RaTG13 in the remainder of the viral genome. Bat CoV RaTG and the human 2019-CoV2 have only 89.2% amino acid similarity in RBD. Indeed, the Guangdong pangolin coronaviruses and 2019-CoV2 possess identical amino acids at the five critical residues of the RBD, whereas RaTG13 only shares one amino acid with 2019-CoV2 (residue 442, human SARS-CoV numbering).
Кстати, авторы этой статьи тоже выделили явную филогенетическую мозаичность шиповидного белка CoV2:
Интересно, что филогенетический анализ только синонимичных сайтов в RBD показал, что филогенетическое положение гуандунского панголина согласуется с положением по остальной части вирусного генома, а именно, что он не является ближайшим родственником 2019-CoV2 (Figure 2b). Следовательно, возможно, что сходство аминокислот в RBD коронавирусов панголина и 2019-CoV2 обусловлено селективно-опосредованной конвергентной эволюцией, а не рекомбинацией, хотя на имеющихся данных трудно выбрать между этими сценариями.
Исходный текст
Interestingly, a phylogenetic analysis of synonymous sites alone in the RBD revealed that the phylogenetic position of the Guangdong pangolin is consistent with that in the remainder of the viral genome, rather than being the closest relative of 2019-CoV2 (Figure 2b). Hence, it is possible that the amino acid similarity between the RBD of the Guangdong pangolin coronaviruses and 2019-CoV2 is due to selectively-mediated convergent evolution rather than recombination, although it is difficult to choose between these scenarios on current data.
В переводе с научного, слова авторов означают, что если проанализировать весь RBD трёх штаммов, отбросив явные различия (несинонимичные замены) между ними, которые, в основном, приходятся на RBM (который, напомню, идентичен между CoV2 и Pangolin), и построить филогенетическое древо по синонимичным заменам, то CoV2 таки ближе к RaTG13, а не к панголиньему штамму. Что довольно странно в свете того, что у панголиньего с CoV2 идентичный RBM (то есть отрезок внутри RBD).
Далее авторы теоретизируют, что это может быть следствием конвергентной эволюции, то есть, иначе говоря, что штаммы CoV2 и панголинов пришли к идентичному RBM каждый своей дорогой, а не через совместную рекомбинацию общих предков. Потому что действительно уж очень странная должна была произойти рекомбинация — как будто кто-то просто взял кусок RBM из панголиньего штамма и заменил им RBM в RaTG13. А это уже смахивает не на эволюцию, а на, прости Дарвин, Intelligent Design.