
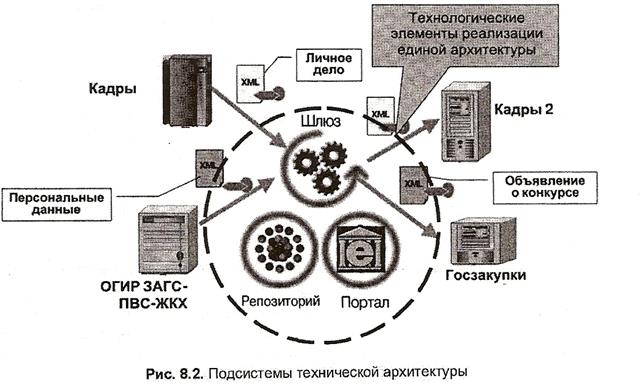
Архитектура электронного правительства: Единая архитектура – это методологический подход при создании системы управления государства, который строится...
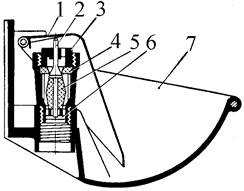
Индивидуальные и групповые автопоилки: для животных. Схемы и конструкции...

Архитектура электронного правительства: Единая архитектура – это методологический подход при создании системы управления государства, который строится...

Индивидуальные и групповые автопоилки: для животных. Схемы и конструкции...
Топ:
Особенности труда и отдыха в условиях низких температур: К работам при низких температурах на открытом воздухе и в не отапливаемых помещениях допускаются лица не моложе 18 лет, прошедшие...
Оценка эффективности инструментов коммуникационной политики: Внешние коммуникации - обмен информацией между организацией и её внешней средой...
Характеристика АТП и сварочно-жестяницкого участка: Транспорт в настоящее время является одной из важнейших отраслей народного...
Интересное:
Подходы к решению темы фильма: Существует три основных типа исторического фильма, имеющих между собой много общего...
Влияние предпринимательской среды на эффективное функционирование предприятия: Предпринимательская среда – это совокупность внешних и внутренних факторов, оказывающих влияние на функционирование фирмы...
Финансовый рынок и его значение в управлении денежными потоками на современном этапе: любому предприятию для расширения производства и увеличения прибыли нужны...
Дисциплины:
|
из
5.00
|
Заказать работу |
|
|
|
|
Чтобы обеспечить параллельное секвенирование 37 фрагментов мтДНК в до 60 образцах на машине 454 GS FLX, штрих-коды были лигированы с мультиплексированными фрагментами ДНК в соответствии с протоколом мечения Meyer 2008 (ссылка 54), за исключением того, что весь продукт ПЦР был использован для начального этапа. Три мультиплексных ПЦР одного образца объединяли во время первой стадии очистки. Для всех стадий очистки использовали набор для очистки ПЦР Qiagen MinElute. 454 Подготовка библиотеки и секвенирование были выполнены GATC Biotec AG в Констанце. Отдельные продукты ПЦР анализировали с использованием анализатора последовательности ABI 3130 после стандартного секвенирования цикла Сэнгера с использованием набора для определения последовательности циклов BigDye Terminator v1.1 (Applied Biosystems, Life Technologies, Дармштадт, Германия). Для ядерных маркеров избыточные последовательности были свернуты с использованием CD-HIT-454 (ссылка 55), отдельные кластеры последовательностей были удалены и уникальные последовательности были сопоставлены с mafft 56к контрольной последовательности для каждого из маркеров и аллелей подсчитывали. Для последовательностей мтДНК консенсусные последовательности были созданы с помощью SeqMan из пакета DNASTAR Lasergene, а гаплогруппы были назначены с помощью веб-приложения HaploGrep, использующего сборку phylotree 15 (ссылки 57, 58) (дополнительные таблицы 4–6).). Чтобы подтвердить подлинность результатов, каждая позиция HVR1 должна была быть покрыта, по крайней мере, тремя однозначными последовательностями из независимых ПЦР. Фрагменты кодирующей области секвенировали по меньшей мере дважды с 454 FLX. Самостоятельное воспроизведение было выполнено для 22 образцов А. Пилипенко в Институте цитологии и генетики СО РАН. Все результаты согласуются с результатами, полученными в лаборатории палеогенетики, Майнц. Во время процесса подготовки образца холостой контроль включали в стадии измельчения, экстракции и амплификации. Общий уровень загрязнения составил 2,2%. Загрязнения контролировали и сравнивали с образцами последовательностей тех же стадий реакции.
|
|
Биостатистический анализ
Для популяционного генетического анализа были использованы последовательности HVR1 из положения 16,040–16,400 из 96 образцов, проанализированных для этого исследования, и были использованы дополнительные 51 образец, взятый из литературы (дополнительная таблица 1). Программное обеспечение 59 Arlequin 3.5.1.3 использовалось для AMOVA и для расчета разнообразия генов (разнесения гаплотипов) 60, разнесения нуклеотидов 60, 61, значений F ST и расстояния Рейнольдса 62. Метод попарного расстояния использовался для расчета значений F ST с 1000 перестановками и значением гаммы = 0. Фу F S тест селективной нейтральности был выполнен для проверки расширения популяции. Уровень значимости для FЗначения S были установлены на уровне 0,02 (ссылка 63). Результаты сводной статистики см. В дополнительной таблице 7.
ABC анализ
Для изучения демографической истории скифов мы сформулировали несколько сценариев-кандидатов, которые послужили основой для моделирования выборок генетических данных для региона HVR-1 с использованием BayeSSC 64, 65. Расчеты сводной статистики по наблюдаемым данным были выполнены в DNaSP v5 (ссылка 66). Чтобы подтвердить, что сценарии-кандидаты смогли воспроизвести наблюдаемые генетические данные, мы сравнили предыдущие распределения смоделированной суммарной статистики с эмпирически наблюдаемыми значениями. Все анализы были выполнены с использованием пакета abc в R 2.15.1 (ссылки 67, 68). Мы ссылаемся на дополнительное примечание 1 для получения полной информации о демографических сценариях и анализах, используемых здесь.
|
|
Геномный анализ
Подготовка библиотеки ДНК для последующего секвенирования ружья проводилась в соответствии с протоколом, использованным в Kircher 2012 (ссылка 69), с небольшими изменениями для образцов ружья (дополнительное примечание 3). Для анализа геномных данных были вычислены f 3- и f 4 -статистики (qp3Pop и qpDstat из ADMIXTOOLS) 37, был применен анализ потоков предков (qpWave / qpAdm) 26 и был выполнен анализ ADMIXTURE 38, 39 (дополнительное примечание 2),
Доступность данных
Файлы BAM для геномных данных можно загрузить из Европейского архива нуклеотидов под регистрационным номером PRJEB18686. Митохондриальные последовательности были депонированы в GenBank под регистрационными номерами KY369766-KY369861.
Дополнительная информация
Как цитировать эту статью: Unterländer, M. et al. Родословная и демография и потомки кочевников железного века Евразийской степи. Туземный Commun. 8, 14615 дои: 10.1038 / ncomms14615 (2017).
Примечание издателя: Springer Nature остается нейтральным в отношении юрисдикционных претензий на опубликованных картах и институциональных принадлежностей.
|
|
|

Историки об Елизавете Петровне: Елизавета попала между двумя встречными культурными течениями, воспитывалась среди новых европейских веяний и преданий...

Археология об основании Рима: Новые раскопки проясняют и такой острый дискуссионный вопрос, как дата самого возникновения Рима...
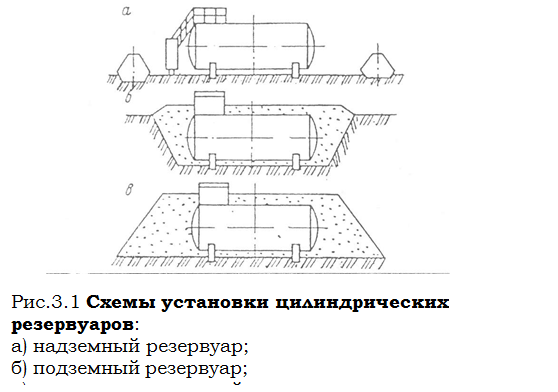
История развития хранилищ для нефти: Первые склады нефти появились в XVII веке. Они представляли собой землянные ямы-амбара глубиной 4…5 м...

Своеобразие русской архитектуры: Основной материал – дерево – быстрота постройки, но недолговечность и необходимость деления...
© cyberpedia.su 2017-2024 - Не является автором материалов. Исключительное право сохранено за автором текста.
Если вы не хотите, чтобы данный материал был у нас на сайте, перейдите по ссылке: Нарушение авторских прав. Мы поможем в написании вашей работы!